文章来源公众号:智药邦 作者:智药邦
2025年7月17日,Science上发表了一篇Prize essay,题为AI to rewire life’s interactome: Structural foundation models help to elucidate and reprogram molecular biology,作者为大奖得主乔卓然。

乔卓然,本科毕业于北京大学,博士毕业于加州理工学院。2023年至2024年间,他在Iambic Therapeutics担任高级机器学习科学家。2025年起,加入Chai Discovery,出任科学家并成为创始团队成员。他的研究聚焦于以物理为驱动的机器学习方法,旨在解决化学与结构生物学中涉及复杂分子体系的问题。
生物分子间的相互作用源于漫长的进化过程,使生命系统能够在不同的空间和时间尺度上实现信号与能量的传递。若能从这些复杂而广泛的相互作用中提取规律与模式,将有望释放出巨大的分子设计与治疗开发潜力。为实现这一目标,乔卓然与同事们借助人工智能与加速计算等先进工具,构建了一个面向结构互作组的“计算显微镜”,将这一科学愿景转化为可计算的问题。
蛋白质与小分子等生物分子之间的相互作用,决定了更高阶的生物结构组织,并驱动着生命的基本功能。为了识别这些相互作用并理解其发生机制,科学家亟需一种能够解析三维结构的“显微镜”,以从分子体系的局部快照中还原出构成交互的原子坐标。尽管X射线晶体学与冷冻电子显微镜等实验方法在结构解析方面极具威力,但由于实验周期长、样本制备复杂,从分离分子快照到获得可分析样本往往需要数月之久。而“计算显微镜”有望打破这一瓶颈,能够直接根据分子的身份信息合成其结构图景。
这一设想在过去半个世纪里一直被视为遥不可及的“登月计划”,直到近年来基于人工智能的蛋白质结构预测取得突破性进展。与传统模拟方法需穷举天文数量级假设不同,AlphaFold2及相关AI结构预测工具能够借助分子进化留下的痕迹,以及从实验解析结构中学习到的模式,实现高效预测。具体而言,这些模型通过整合进化约束信息(如多序列比对或蛋白质语言模型),并由专门设计的神经网络进行解析,从而以前所未有的准确度推理氨基酸在三维空间中的排布。在此基础上,作者进一步开发了生成式机器学习方法,用以解决两个关键问题:蛋白质–配体相互作用及其构象空间。
生物分子具有高度动态性,需要大量“快照”才能全面捕捉其行为特征。蛋白质的构象常受小分子配体和翻译后修饰的调控,这些因素驱动的构象变化在生物功能调节中发挥关键作用,也为药物发现提供了重要契机。这一复杂性对传统“锁钥模型”式的蛋白–配体相互作用预测方法构成挑战,后者通常假设蛋白质为刚性结构。尽管分子动力学等方法可模拟结合过程及构象变化,但因跨越低能构象状态之间的缓慢跃迁所需计算资源极高,效率受到显著限制。
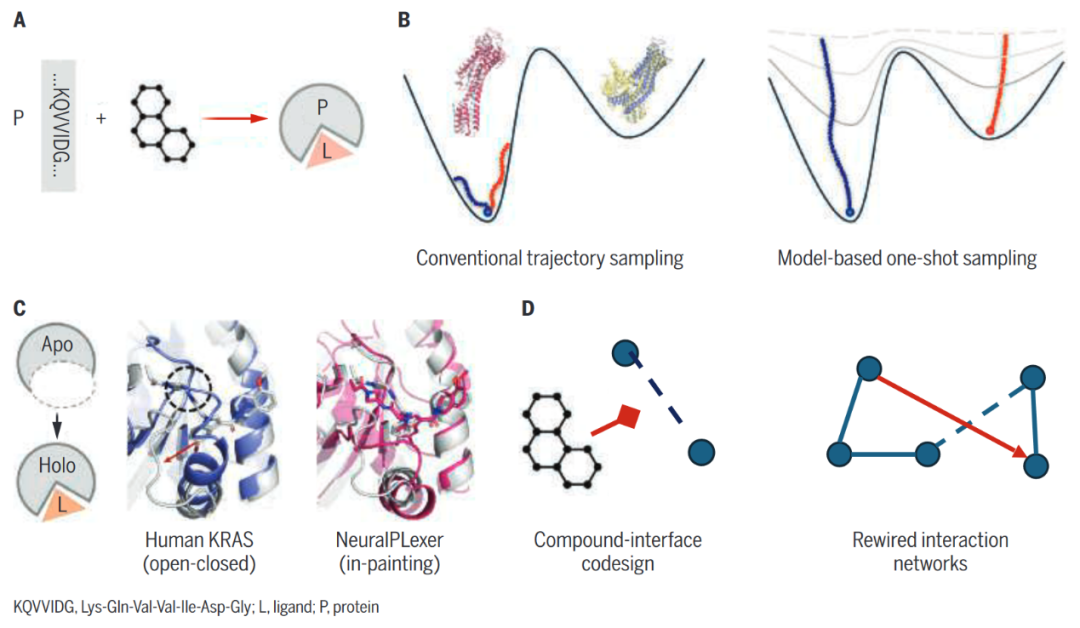
图1 AI在多种模态下统一生物分子结构预测与设计方面展现潜力
(A)蛋白-配体复合物的生成式结构预测示意图。
(B)生成建模可实现分子构象空间的快速采样。
(C)以NeuralPLexer对人类KRAS G12C配体结合状态的预测为例,展示其揭示的结构性见解。
(D)结构预测基础模型与从头分子设计方法所带来的新机遇。
作者提出了一种生成式人工智能策略NeuralPLexer,以解决这一难题(见图)。NeuralPLexer通过生成扩散机制建模蛋白-配体结合的构象景观:从整个分子复合物的初步草图出发,逐步细化所推理出的结构细节。该模型通过对初始草图的多种并行猜测,生成一组构象“快照”,从而覆盖生物分子的热力学构象空间。这种“一步生成”的方式提供了一种跳过传统采样瓶颈的路径,使得能够快速获得分子相互作用的全貌,并具有原子级别的细节。NeuralPLexer所采用的神经网络结构,模仿了生物分子复合物的多尺度分层组织。它先通过推断的蛋白-配体接触初始化预测,随后生成详细的几何结构表示,同时保持基本的物理对称性。
作为初步验证,作者将NeuralPLexer应用于预测隐蔽结合位点的形成,这类位点仅在配体结合后才出现,在游离状态的蛋白结构中并不存在。通过使用一个涵盖因小分子结合而引发显著构象变化的数据集,NeuralPLexer成功生成了与实验解析结果一致的构象分布。在一组多样的酶系统中,其采样得到的构象集合与实验解析的蛋白质构象高度一致,相关性可通过TM-score和Q因子等指标量化,从而有效克服了静态蛋白质折叠模型的局限性。此外,NeuralPLexer还能为其生成的构象预测分配置信度评分,展现出区分强亲和配体与弱亲和配体的能力,且这一性能是在未曾使用结合亲和力数据进行训练的情况下实现的,并可适用于广泛的靶点类型。
作者进一步将该策略用于解析蛋白质功能的分子机制。例如,在一种酮酸还原异构酶中,其催化机制近来才被揭示,NeuralPLexer能够准确捕捉到在辅因子和抑制剂结合时N-亚结构域的闭合运动,并提供了关于该靶点自组装行为的线索。在另一例GPCR中,模型生成的构象假设成功解释了该受体在无配体状态下仍具备的构成性活性。这些结果凸显出NeuralPLexer在研究别构调控与酶催化等关键分子机制中的强大潜力。
除了原子级构象之外,作者团队还开发了几何深度学习方法OrbNet-Equi,用于研究分子相互作用的能量特征。该方法在主族化学体系中达到了可与全精度量子力学方法相媲美的准确度,同时计算速度提升了约1000倍。通过整合这些工具,能够以更高分辨率解析质子化状态的变化,以及电子电荷与自旋构型,从而提供一种全面的策略,用于解析和设计质子或电子转移路径。
除了对已知蛋白-小分子相互作用的结构预测外,作者团队还通过引入一种“补图(inpainting)”策略,拓展了NeuralPLexer的功能,用以发现新的结合口袋。这一方法有望帮助为先前尚未表征的结合位点设计配体。类似于AI图像编辑工具根据周围内容填充并重绘选定区域,该方法仅凭靶蛋白的主链信息,就能同时推断配体结构、蛋白序列和结合口袋形状的变化。在诸如 KRAS-G12C 等具有挑战性的靶点上,该方法的设计成功率显著优于传统构象搜索和分子对接算法。
随着结构预测基础模型准确度的不断提升,未来的研究将进一步拓展其适用范围,以覆盖更复杂的相互作用模式和任意计量比的生物组装体。这包括通过化学调节剂稳定的蛋白-蛋白相互作用界面,即所谓的诱导接近(induced proximity)现象。这类技术进展将为重新编程蛋白相互作用网络、恢复健康细胞状态的配体理性设计开辟道路。统一的相互作用预测模型与从头设计结合配体的框架结合,将成为一个强大的平台,可用于设计稳定蛋白-蛋白界面的调节剂,调控致病蛋白的亚细胞定位,并偏向性地促进特定组装状态的形成。这种“重构细胞信号通路”的方法不仅有望提升治疗的精准性,还可通过选择性靶向蛋白的寡聚状态、保留对正常细胞功能至关重要的单体形式,从而扩大治疗窗口。上述新能力有望推动结构基础模型向多功能工具演进,促进化学生物学研究,并加速新药的发现进程。